コーヒーブレイク
GC-richサンプルのPCRバイアスを調べてみた!
■はじめに
NGSのライブラリー増幅の際、GC-richやAT-richなサンプルが効率的に増幅されなかったことはありませんか?
均一な増幅ができていない場合、シークエンス結果が不正確になってしまったり、シークエンスの定量性が失われてしまったりする可能性があります。また必要なシークエンス情報を得るため、より多くの配列データ(リード)が必要となり、よりコストがかかることにもなります。NGSのライブラリー増幅には均一な増幅ができるPCR酵素を選ぶことが非常に重要です。
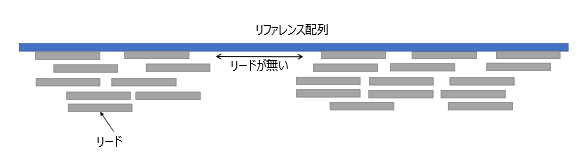
既知の配列をNGSで解析する場合、得られたリードをリファレンス配列と照合(マッピング)します。一度で膨大な量のリードが得られるため、リファレンス配列上にさまざまなリードが重なり合うようにマッピングされます。
しかし、ライブラリー増幅の際、偏りが生じてしまうと、特定の領域のリードが得られにくくなり、シークエンス結果が不正確になってしまいます。
今回は、GC-richなライブラリーを増幅して、PCR酵素の違いでシークエンスデータにどれくらい差が出るのか試してみました。
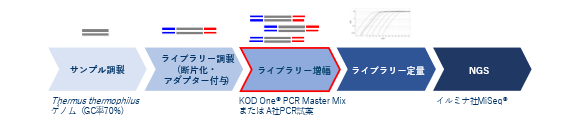
■実験方法
GC率70%のThermus thermophilusゲノムを断片化し、イルミナ社のP5、P7配列を持つアダプターを付加しました(ライブラリー調製)。KOD One® PCR Master Mix とA社PCR試薬を用いて上記ライブラリーを増幅した後、イルミナ社 MiSeq®を使用してシークエンスを行い、データを比較しました。(A社の反応液組成、PCRサイクルはA社推奨条件で実施しました。)
滅菌蒸留水 14μL
KOD One® PCR Master Mix 25μL
10μM Forward Primer 5μL
10μM Reverse Primer 5μL
1ng/μL NGSライブラリー 1μL
Total Volume 50μL
PCR 変性 98℃ 10秒 → アニーリング 60℃ 5秒 → 伸長 68℃ 1秒 (14 cycles)
【 Primer配列 (TruSeq用プライマー) 】
Fwd: AATGATACGGCGACCACCGAGATC
Rev: CAAGCAGAAGACGGCATACGAG
■結果
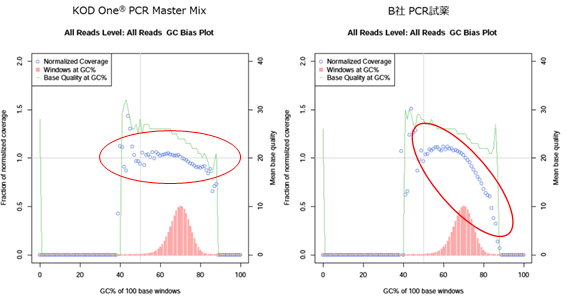
以下の図は、シークエンスされたリードのGCバイアスを比較したものです。
X軸のライブラリーのGC率に対して、Y軸のノーマライズされたカバレッジ(下図:〇)が1に近いほど、バイアスなくライブラリーが増幅したことを意味します。KOD One® PCR Master Mixで増幅したライブラリーはGC率が高くても低バイアスであることが示されました。
また、同じリード数でリファレンス配列におけるカバー率を比較した結果もKOD One® PCR Master Mixで増幅したライブラリーの方が高いカバー率を示しました。
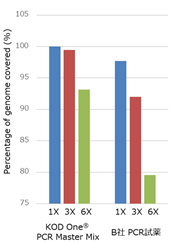
1X:リファレンス配列におけるリード数が1つ以上のカバー率
3X:リファレンス配列におけるリード数が3つ以上のカバー率
6X:リファレンス配列におけるリード数が6つ以上のカバー率
KOD One® PCR Master Mixは長鎖ターゲットでも均一かつ高速に増幅が可能です。Shortリードのライブラリーだけでなく、Longリードのライブラリー増幅にも応用が期待できます!
サンプル種、鋳型のサイズ分布によって、最適なサイクル数は1~3サイクル前後する場合がありますが、ライブラリーのインプット量とサイクル数に関しては、以下をご参照ください。
| インプット量 | サイクル数 |
| 1μg | 0-1 |
| 500ng | 1-2 |
| 100ng | 4-5 |
| 50ng | 5-6 |
| 10ng | 8-10 |
| 1ng | 13-15 |
| 0.25ng | 16-18 |
■さいごに
今回使用した製品はこちら
KOD One® PCR Master Mixシリーズ

高い正確性・増幅効率をもつ改変型KOD DNA polymeraseを用いたPCR用2×マスターミックスです。
優れた正確性と簡便さを持ち、高速PCRが可能です。
KOD One® PCR Master Mixシリーズの他の実施例はこちら
その他の製品の実施例はこちら







