コーヒーブレイク
SNP解析でお酒の代謝に関する遺伝子を調べてみた!
■はじめに
アルコール(エタノール)はまず、肝臓内でアルコール脱水素酵素(ADH)などによって有害物質のアセトアルデヒドに分解されます。その後、アルデヒド脱水素酵素(ALDH)によって酢酸に分解され、最終的には、酢酸は全身に送られ、二酸化炭素と水に分解され、体外へ排出されます。
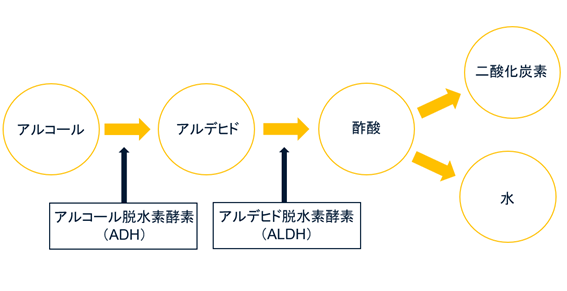
二日酔いの原因となる有害物質アセトアルデヒドを分解する酵素の1つがALDH2であり、この遺伝子の違いがお酒の強いか弱いかに関わるといわれています。
このALDH2の504番目のアミノ酸がグルタミン酸の場合(112241766番目の塩基が「G(グアニン)」の場合)、アセトアルデヒドを酢酸に分解する活性が高く、リジン(Lys)の場合(112241766番目の塩基が「A(アデニン)」の場合)はその活性が無くなります。そのため、Glu/Gluの人はお酒が強いのに対し、Lys/Lysの人はほとんどお酒が飲めず、Glu/Lysの人はある程度はお酒が飲めますが、顔が赤くなりやすい傾向があります。
そこで、今回は、身近な遺伝子であるお酒の代謝に関する遺伝子ALDH2について一塩基多型(SNP)を調べてみました。
果たして研究員たちの結果は…?
■実験方法
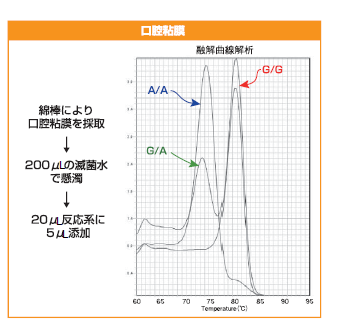
綿棒で研究員の口腔粘膜を採取し* 200μLの滅菌水で懸濁したのち、KOD SYBR® qPCR Mixを使用してASP(Allele specific primer)-PCRを用いるALDH2のSNP解析を融解曲線解析で行いました。
*:研究員からの検体採取にあたり同意を取得しております。
滅菌蒸留水 3.4μL
KOD SYBR® qPCR Mix 10μL
10μM 共通Primer 0.4μL
10μM G特異的Primer 0.4μL
10μM A特異的Primer 0.4μL
50×ROX referencre dye 0.04μL
DNAサンプル 5μL
Total Volume 20μL
(ABI7500 Fast使用)
98℃, 2min.
↓
98℃, 10sec.
60℃, 10sec.
68℃, 30sec.
35 cycles
↓
Melting Curve
(プライマー)
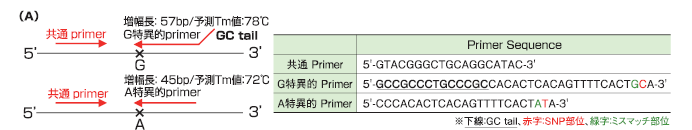
ワンチューブで遺伝子型の識別を可能にするために、一方のアレル特異的プライマーにGC tail(下線部)を付加し、増幅産物の融解温度(Tm値)に差異が生じるように設計しました。GC tail付加による融解温度の上昇を大きくするため、増幅長を100bp以下にしています。また、この実施例においては、アレル特異的プライマーとして3’側から2塩基目をSNP部位(赤)、3塩基目をミスマッチ(緑)になるように設計しました。
■結果
研究員3人のうち、1人はG/G(ALDH2活性型遺伝子=お酒に強い)、1人はG/A(低活性型遺伝子=顔は赤くなるがある程度は飲める)、1人はA/A(失活型遺伝子=ほとんど全く飲めない)という結果になりました。

■さいごに
今回ご紹介したASP-PCRは、一方のプライマーの5’末端にGC tailを付加することにより、同サイズのターゲットの識別を融解曲線解析により行うことができます。さまざまな原理に応用可能です。
いずれにせよ、お酒は節度ある適量(飲めない方は少量でも危険です!)で楽しみましょう。
今回使用した製品はこちら
KOD SYBR® qPCR Mix
本製品はSYBR® Green I検出系によるリアルタイムPCR用 2×濃度のマスターミックス試薬です。
3’→5’エキソヌクレアーゼ活性(校正活性)を除去したKOD exo(-) DNA polymeraseと最適化されたバッファー組成を組み合わせることで、KODの優れた合成能やクルード成分の阻害を受けにくいという性質を最大限に発揮させることを可能としました。