実施例
KOD FX 実施例26 DGGE法を用いた沿岸海域に生息する植物プランクトン(クリプト藻)の多様性の解析
【遺伝子名】PsbA(光化学系II反応中心のD1タンパク ) 特にクリプト藻をターゲットとした
【サンプル】海水の(デュラポア)フィルターろ過サンプル
【サンプル調製方法】
ろ過したフィルターに溶解液*1mLを加え、55℃で1時間処理
→フィルターを除去し、遠心(10,000×g, 10分)処理
→上清1~1.5μLをテンプレートとして使用
*溶解液組成:20mM Tris-HCl(pH8.0), 5mM EDTA, 0.3% SDS, Proteinase K 200μg/mL
【ターゲット長】約350bp
【プライマー配列】
Primer F:5'-CGCCCGCCGCGCCCCGCGCCCGTCCCGCCGCCCCCGCCCCATGCGTCCTTGGATTTCTGT-3' (60mer)
Primer R:5'-CCARATACCTACAACTGGCCATA-3' (23mer)
【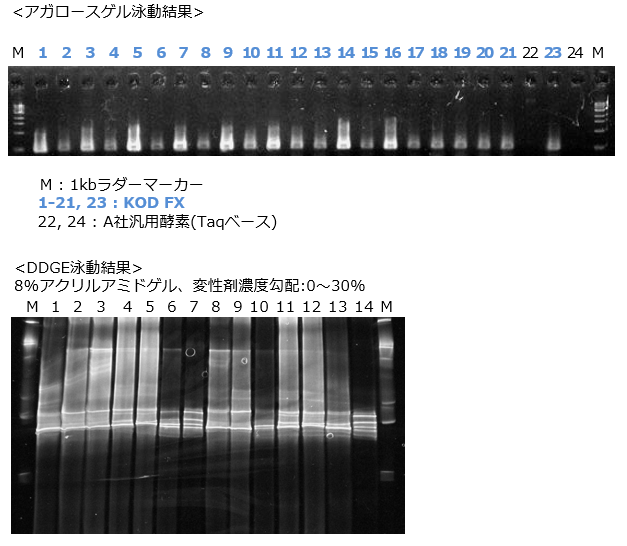 PCR条件】
PCR条件】
PCR grade water 3.3μL
2×PCR buffer for KOD FX 15μL
2mM dNTPs 6μL
Primer F (5pmol/μl) 1.8μL
Primer R (5pmol/μl) 1.8μL
KOD FX (1.0U/ ml) 0.6μL
サンプル 1.5μL
---------------------------------------------------
Total volume 30μL
【PCRサイクル】
94℃ 2 min.
(98℃ 10 sec., 55℃ 30 sec., 68℃ 10 sec.)×35cycles
他社製品は推奨条件で行いました。
【データご提供】
(独)水産総合研究センター 東北区水産研究所
奥村 裕先生
【先生からのコメント】
通常A社汎用酵素(Taqベース)でPCRがうまくいかないことはわかっていたので、シーケンスの際は、 A社汎用酵素 + B社クルードサンプル対応PCR添加剤を使用した後TAクローニングを行っていました。ただ、 PCR添加剤は粘性が高く、アガロースゲルを使って確認する際は問題はないのですが、PCR産物を精製せずにDGGEに使用するとバンドがスメアになり、ゲル濃度や変性剤の濃度を変えてもうまく電気泳動できませんでした。
KOD FXを用いた場合は、DNAの抽出を行わなくてもPCRが可能で、しかもDGGEも問題なく行えました。ホームページを拝見すると、ProK、SDSを使用したサンプルは精製が必要と書かれていましたが、すでに加えて処理したテンプレートしかなかったので、そのままPCRを行いましたが問題なくPCRできました。サイクル数に関しては最初30サイクルで行ってみましたが、35サイクルの方が良かったです。また、GCクランプのついたプライマーで、うまくいくか心配だったのですが、その点も問題なくPCRでき助かりました。
 KOD FX 製品ページ
KOD FX 製品ページ
 実施例一覧ページ
実施例一覧ページ







