実施例
Blend Taq 実施例5 ランダム配列の増幅
PCR用酵素を利用して核酸配列の増幅を行う場合、増幅されやすい配列と増幅されにくい(あるいはされない)配列があります。増幅する配列の“好き嫌い”の少ない酵素を使用することで、未知配列のクローニング成功率を高めたり、PCR条件の統一化が容易になります。今回はBlend Taq® -Plus-によるランダム配列の増幅についてご紹介します。
【方法】
1.PCR
センスプライマー(SP):5'Texas Red標識24merプライマー
アンチセンスプライマー(AP):非標識24merプライマー
鋳型DNA:Total 98mer合成核酸 (SP相同配列)-(ランダム配列[50 mer])-(AP相補配列)

2. 増幅反応生成物の解析
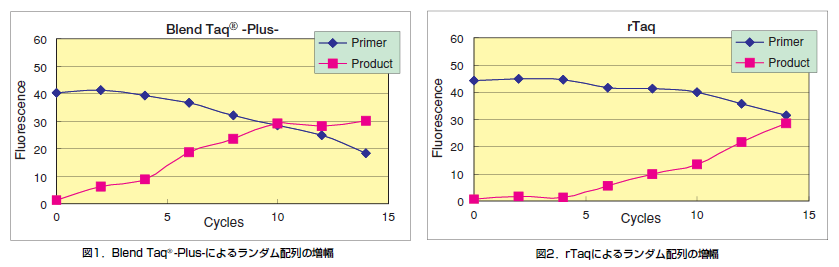
10%ポリアクリルアミドゲル電気泳動後、染色せずにUVサンプル撮影装置「FAS-Ⅲ」による画像撮影を行いました。得られた画像から画像解析ソフトにより増幅産物(Product)及び未反応蛍光プライマー(Primer)の定量化を行いました。
【結果】
50merのランダム配列は理論上450=1030通りとなりますが、実際にPCR反応の鋳型として供されたのは50pmolesであるため、約3×1013種類の配列が一分子ずつ入っている計算になります。
Blend Taq® -Plus-では2サイクルで増幅産物が確認でき(図1)、10サイクルで増幅産物と未反応プライマーがほぼ等量になりました。一方、rTaqではそれぞれ4サイクル遅い、6サイクルと14サイクルでした(図2)。したがって、Blend Taq® -Plus-はrTaqと比較して理論値で16(= 24)倍多くの種類の配列を鋳型として利用できたことがわかりました。
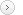 Blend Taq 製品ページ
Blend Taq 製品ページ
 実施例一覧ページ
実施例一覧ページ







