実施例
ReverTra Ace qPCR RT Master Mix 実施例5 cDNA特異的検出例 -夾雑ゲノムDNA除去性能の確認-
リアルタイムPCRによる遺伝子発現解析実験では、cDNAのみを検出することが重要です。多くの場合、エキソンジャンクションを挟む位置にプライマーを設計することで、鋳型RNAに混入したゲノムDNA(gDNA)に由来する増幅を回避することができます。
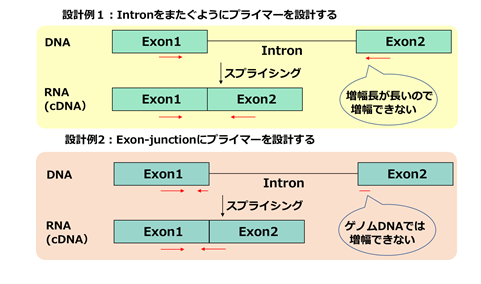 しかしながら、cDNA特異的なプライマーが設計できないためにgDNAに由来する増幅が起こり、特に低発現遺伝子の研究などにおいて、発現量が正確に解析できない場合があります。
しかしながら、cDNA特異的なプライマーが設計できないためにgDNAに由来する増幅が起こり、特に低発現遺伝子の研究などにおいて、発現量が正確に解析できない場合があります。
< cDNA特異的Primerが設計できないケース >
- ターゲットにイントロンがない(intronless遺伝子)か、あるいは、イントロンがあってもサイズが短く、十分な大きさのイントロンをまたぐ位置にプライマーを設定できない場合
- ターゲットに偽遺伝子(Pseudogene)が存在する場合
ReverTra Ace® qPCR RT Master Mix with gDNA Removerには、強力なDNA分解活性を有するgDNA Removerが付属されており、逆転写反応に先立って本試薬で鋳型RNAを処理することによって、ゲノムDNAフリーのcDNAを簡便に合成することができます。本キットを用いてcDNA特異的検出を行った例をご紹介いたします。
【実験方法】
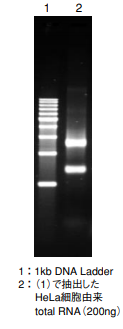 Total RNAの抽出
Total RNAの抽出
まず、市販のスピンカラムタイプのtotal RNA抽出キットを用いて、HeLa細胞からTotal RNAの抽出・精製をおこないました(カラム上でのDNase処理はなし)。抽出後、RNAの品質を確認するため、1%アガロースゲルを用いて電気泳動を行いました(右写真)。
- cDNA合成(逆転写反応)
次に、本試薬を用いて、抽出したHeLa Total RNA 500ngを、逆転写酵素の有り(+)無し(-)の条件で逆転写反応(20μL反応系)を行いました。その際、逆転写酵素無し(-)の条件には、キット付属のno-RT Controlを用いました。また、比較のため、gDNA除去機能を持たないcDNA合成キットを用いて、同様に逆転写酵素の有り(+)無し(-)の条件にて逆転写反応を行いました。
- リアルタイムPCR
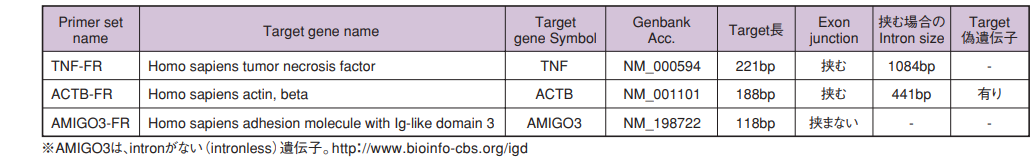
続いて、以下のリアルタイムPCR条件にて、TNF, ACTB, AMIGO3の各遺伝子の検出を行いました(N=2)。
試薬:THUNDERBIRD® SYBR™ qPCR Mix [Code No.QPS-201]
鋳型:2で調製した反応液2μL/20μL反応系(持込量10%)
測定:Applied Biosystems® 7900HT
各遺伝子の検出には、以下のプライマーセットを使用しました。
【結果および考察】
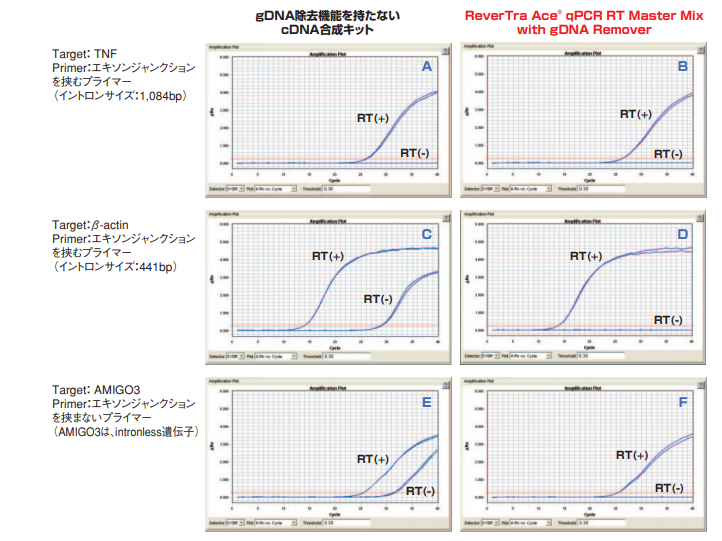
イントロンが十分に長いTNF (図A) では、RT(-)条件で増幅はみられませんでしたが、偽遺伝子が存在するハウスキーピング遺伝子β-actin (図C) やイントロンを挟まなかったAMIGO3 (図E) においては、RT(-)条件でも増幅がみられました。β-actinでは融解曲線解析の結果から、偽遺伝子由来の産物が優先的に増幅している可能性が示唆されました。このことから、gDNA除去機能をもたないcDNA合成キットの場合、プライマーセット(あるいはTarget)によっては、RNAサンプルに夾雑しているgDNAに由来するシグナルが検出されてしまう可能性があると考えられます。
これに対し、ReverTra Ace® qPCR RT Master Mix with gDNA Removerでは、いずれの場合においても、RT(-)条件で増幅がみられず (図B, D, F)、確実にgDNAが除去され、gDNAに由来するシグナルは検出されないことが確認されました。
以上より、混入gDNA由来の増幅は、本キットを使用することにより、回避することが可能でした。この効果は、低発現遺伝子の発現解析を行う場合に特に有効であると考えられます。
 ReverTra Ace® qPCR RT Master Mix 製品ページ
ReverTra Ace® qPCR RT Master Mix 製品ページ
 実施例一覧ページ
実施例一覧ページ







