コーヒーブレイク
さまざまなRNA量からRNA-seqをしてみた!
■はじめに
RNAがたくさん取れた時、インプット量はどれくらいにするべきか迷ったことはありませんか?
GenNext® Shin-RamDA-seq® Single Cell Stranded Kitでは推奨のインプット量の範囲は10pg~1ngとなっています。
インプット量の違いでどのようなライブラリーが得られるのか、検出遺伝子数がどうなるのか、今回、GenNext® Shin-RamDA-seq® Single Cell Stranded Kitを用いてさまざまなインプット量からRNA-seqを実施してみました。
■実験方法
ヒト白血病細胞株K562細胞から抽出したRNAを用いて、インプット量を10 pg~31.25 ngの範囲でライブラリー調製を行いました。
cDNA合成には、NSR Primer Set for human [Code No. NSR-101]を使用し、ライブラリーエンリッチメントはすべて19サイクルで行いました。
シーケンスにはイルミナ社 MiSeq®を用い、NGS解析にはbasespace (illumina)のRNA-Seq Alignment (ver. 2.0.2)、 またはRamDAQ (ver.1.9)を使用しました。
■結果
<ライブラリー泳動図>
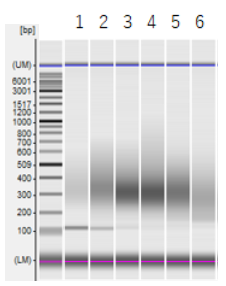
| 1 | 2 | 3 | 4 | 5 | 6 | |
| input RNA 量 | 10pg | 50pg | 250pg | 1.25ng | 6.25ng | 31.25ng |
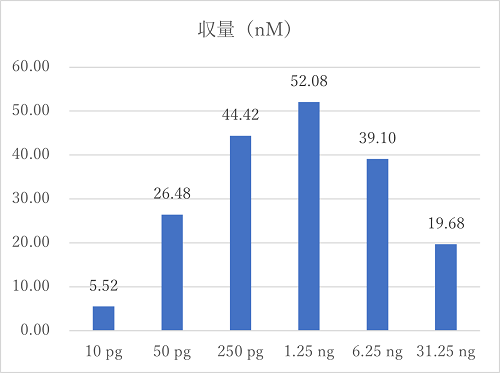
| 収量(nM) | 5.52 | 26.48 | 44.42 | 52.08 | 39.10 | 19.68 |
| 平均鎖長(bp) | 354 | 357 | 332 | 334 | 320 | 286 |
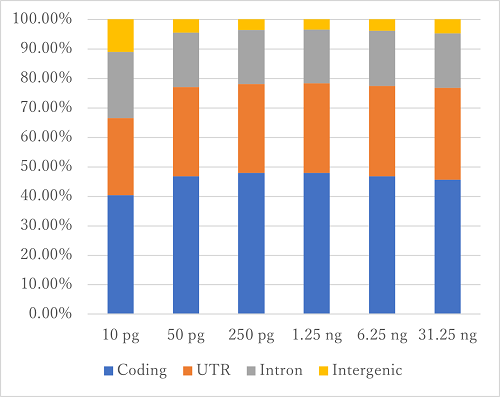
| 収量(nM) | 検出遺伝子数 (Genes) |
検出転写物数 (Transcripts) |
%マッピング | rRNA率(%) | |
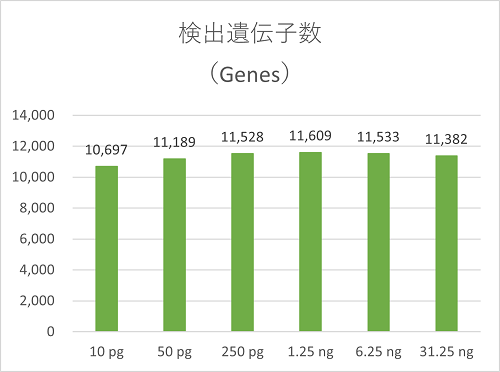
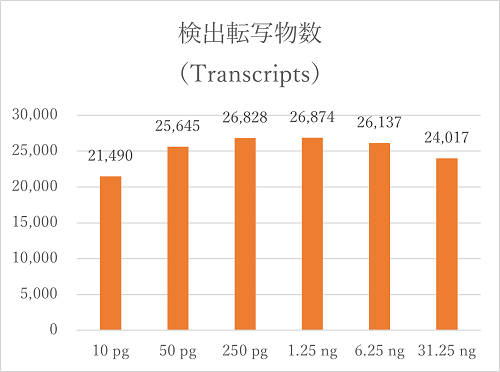
| 10pg | 5.52 | 10,697 | 21,490 | 74.94% | 17.56% |
| 50pg | 26.48 | 11,189 | 25,645 | 90.35% | 19.27% |
| 250pg | 44.42 | 11,528 | 26,828 | 92.04% | 20.84% |
| 1.25ng | 52.08 | 11,609 | 26,874 | 91.17% | 17.93% |
| 6.25ng | 39.10 | 11,533 | 26,137 | 90.04% | 17.51% |
| 31.25ng | 19.68 | 11,382 | 24,017 | 86.94% | 10.95% |
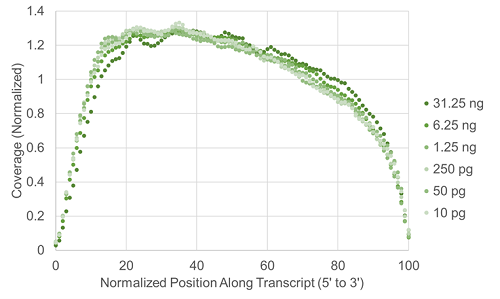
今回、インプット量が6.25 ngの場合でも、高い検出転写産物・遺伝子数を示し、 カバレッジにおいても、偏りのない結果が得られました。
一方で、インプット量が多すぎると平均鎖長が短くなり、ライブラリー収量も減少する傾向が見られました。
RNAが多量にある場合も、GenNext® Shin-RamDA-seq® Single Cell Stranded Kitを用いる場合は1ng程度を添加するのが最も優れたパフォーマンスが得られるようです。
これらの結果は用いるサンプルによっても異なりますので、あくまで参考としてお楽しみください。
■さいごに
今回使用した製品をご紹介します。
RT-RamDA®法による NGS解析用ライブラリー調製キット
GenNext® Shin-RamDA-seq® Single Cell Stranded Kit

●ストランド情報を維持しより正確な解析が可能
アンチセンスやオーバーラップ遺伝⼦を明確に⾒つけることができます。
●シングルセルや微量RNAに対応
1〜100細胞または10pg〜1ng total RNAからcDNAの調製が可能です。
●Poly(A) RNAだけでなくRNAの解析が可能
non-poly(A)RNA (lncRNA,pre-mRNA, circRNAなど)や分解したRNAにも使⽤できます。
●RNA全⻑をカバーした解析が可能
⻑鎖RNAやスプライシングバリアントが正確に解析できます。
*「Shin-RamDA-seq」と「RT-RamDA」 は国立研究開発法人理化学研究所の登録商標です。
*「GenNext」は東洋紡株式会社のNGS関連試薬の登録商標です。
*記載している会社名および・ロゴマークなどは、各社の商号、商標または登録商標です。







