実施例
KOD FX 実施例34 淡水産シオグサ属藻類(緑藻)のDNA断片増幅
【背景】
現在、当研究室では汽水湖におけるシオグサ属藻類(緑藻)の多様性の研究を行っている。その一環として、天然藻体を用いたダイレクトシークエンスを行っているが、その際にはPCRによるDNAの増幅が不可欠である。サンプルとして用いているシオグサ属藻類は多糖類を多く含むことが知られており、これらはPCRにおいてDNAポリメラーゼの酵素反応を阻害する可能性があると考えられている。そのため、これまではテンプレートとなるDNA抽出の際には多糖およびタンパク質を比較的効率よく除去できるとされるCTAB法を用い、PCRはA社高効率Taqを用いてDNAの増幅を行ってきた。しかし、高塩濃度域から採集してきた個体に関しては問題なく増幅が見られたものの、低塩濃度および淡水域から採集してきた個体に関してはこれまでの方法ではPCRによる増幅が殆ど見られなかった。そのため、CTAB法以外のDNA抽出、フェノール / クロロホルム法等によるDNAの精製、nestedPCRおよびプライマーの配列や組み合わせに関する試行錯誤を繰り返してきたがこれといった成果は上がらなかった。そこで、本実験では高い正確性と増幅率を持つとされるKOD FXを用いて、淡水域に生育するシオグサ属藻類のDNA断片の増幅およびこれまで増幅の見られなかった原因の模索を目的とした。
【サンプル】淡水産シオグサ属藻類
【サンプルの処理】抽出:CTAB法
【遺伝子名】rRNA遺伝子のITS領域
【ターゲット長】約700~1200bp
【反応液組成】
|
A社高効率Taq |
KOD FX |
||
|
滅菌水 |
34.8 μL |
滅菌水 |
9.0 μL |
|
10×buffer |
5.0 μL |
2×buffer |
25.0 μL |
|
2.5 mM dNTP |
4.0 μL |
2 mM dNTP |
10.0 μL |
|
プライマー(10μM) |
各2.0 μL |
プライマー(10μM) |
各1.5 μL |
|
高効率Taq |
0.2 μL |
KOD FX |
1.0 μL |
|
DNA溶液 |
2.0 μL |
DNA溶液 |
2.0 μL |
【PCRサイクル】
・A社高効率Taq
94℃,2 min.
↓
[ 94℃, 30 sec. 63 or 66℃, 30 sec. 72℃,1 min. ] 30 cycle
↓
72℃,5 min.
・ KOD FX
94℃,2min.
↓
[ 98℃,10 sec. 65℃, 30 sec. 68℃, 2 min. ] 30 cycle
【プライマーの設定位置】
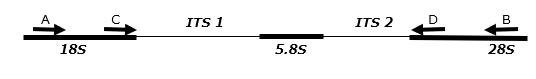
PCRの際に用いたプライマーの位置情報を示す。1回目のPCRの際にはA,Bのプライマー対を用い、nested PCRの際にはC,Dのプライマー対を用いた。
Primer A: 29 mer, Primer B: 23 mer, Primer C: 22 mer, Primer D: 20 mer。
【結果】
今回は4個体に対して、A社高効率Taq・プライマーA,Bを用いたPCR(アニーリング温度63,66℃)、アニーリング温度66℃で行ったPCR産物をテンプレートにして、A社高効率Taq・プライマーC,Dを用いたnested PCR(アニーリング温度63,66℃)およびKOD FX・プライマーA,Bを用いたPCRを行い、計20サンプル(4個体×5通り)の実験を行った。(Table.1)
その結果、A社高効率Taqを用いた通常のPCRでは増幅が殆ど観察されず(Fig. 1 ①,②)、nested PCRでは全体的にスメアとなり、目的の断片の増幅が見られなかった(Fig. 1 ③,④)。しかし、KOD FXを用いたPCRでは目的の断片の増幅が見られた(Fig. 1 ⑤)。
Table 1 PCRの条件
5通りの条件でPCRを行い、各条件のPCR産物に番号(①~⑥)を振った。⑥は過去にA社高効率Taqで増幅の見られたDNA抽出液を用いたポジティブコントロール。
|
テンプレート |
ポリメラーゼ |
プライマー |
アニーリング |
PCR産物 |
|
DNA抽出液 |
A社高効率Taq |
A, B |
63℃ |
① |
|
DNA抽出液 |
A社高効率Taq |
A, B |
66℃ |
② |
|
② |
A社高効率Taq |
C, D |
63℃ |
③ |
|
② |
A社高効率Taq |
C, D |
66℃ |
④ |
|
DNA抽出液 |
KOD FX |
A, B |
65℃ |
⑤ |
|
DNA抽出液 |
KOD FX |
A, B及びC, D |
65℃ |
⑥ |
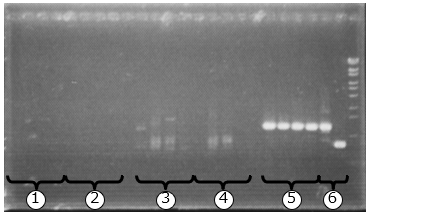 Fig. 1 PCR産物の泳動結果
Fig. 1 PCR産物の泳動結果
PCR産物は各レーンに2.5μLずつ流した。図中の①~⑥はPCRの条件を示し(Table 1) 、 ①,② はA社高効率Taqを用いた通常のPCR 、③,④は①のPCR産物をテンプレートにしたnested PCR、⑤はKOD FXを用いたPCR、⑥は過去にA社高効率Taqで増幅の見られたサンプルのDNA抽出液を用いたポジティブコントロールを示す。
【考察】
今回の実験から、KOD FXは今まで増幅の見られなかった淡水産シオグサのDNA増幅に効果があることが示された。シークエンシングの結果、今まで低塩濃度・淡水域から採集された個体には2種のシオグサ属藻類が含まれていることが判明し、そのうち淡水産として記載されている1種の個体においてのみPCRによる増幅が見られなかったことが判明した。また、海産の種に関しては今までに問題なくPCRでの増幅が見られていたことから、淡水産種と海産種という分布域の違いにより体内に含まれる何らかの物質の有無もしくは量の差があり、それによって増幅の可否が決まっていた可能性が考えられた。KOD FXを用いるまでは、低塩濃度域特有の微細生物のコンタミ・プライマー配列が一致していない等の問題も考えられたが、今回の結果から前者はKOD FXを用いる際には問題ないと判断され、後者に関しては棄却された。また、後日パンフレットに「植物(葉、精米)ライセートからの増幅例」として紹介されていたワンステップ法を用いたPCRも行ってみたが、こちらは増幅が見られたものと見られないものがあった。(データ未掲載)。そのため、ワンステップ法に関してはサンプル数を増やし、もう少し試行錯誤を行う必要があると考えられる。
以上のことから、不純物がかなり多い場合には増幅に支障が出るが、ある程度の純度があればA社高効率Taqと比べると非常に高い増幅効率を示すことが示唆された。
【データご提供】
福井県立大学 海洋生物資源学部
神谷充伸 先生
 KOD FX 製品ページ
KOD FX 製品ページ
 実施例一覧ページ
実施例一覧ページ







