実施例
KOD One 実施例21 次世代シーケンサー(NGS)を用いたSARS-CoV-2亜系統の分類
データご提供
塩野義製薬株式会社 ワクチン開発研究所 | ワクチン基盤研究1グループ
黒岩 美帆 様
<実験の目的>
NGSを用いた、下水に含まれるSARS-CoV-2亜系統を正確な組成比で検出するための技術開発。その精度確認のため、最大7種類のSARS-CoV-2亜系統由来RNAを混合し、正確な組成比で検出できることを実験的に確認した。
(KOD One®はcDNAの増幅・イルミナのオーバーハング配列付与のためNested PCRに使用)
<実験方法>
【サンプルの種類】
SARS-CoV-2のRNA
【鋳型の調製方法】
既知の割合でSARS-CoV-2亜系統のRNAを混合し、混合RNA 1,000コピーをReliance Select cDNA Synthesis Kit(バイオラッド社)にて、下記S008プライマーでcDNA合成したものを鋳型に用いた。
【ターゲット遺伝子名と長さ】
1st PCR : SARS-CoV-2スパイクタンパクのRBD領域(329-535aa)
2nd PCR : SARS-CoV-2スパイクタンパクのRBD領域(337-504aa)
【プライマー配列】
1st PCR
S012: CAACCAACAGAATCTATTGTTAG
S008:AGTTGAAATTGACACATTTG
2nd PCR
S013:TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGTTCCTAATATTACAAACTTGTGC
S009:GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGACTACYACTCTRTATGGTTGGT
【PCR条件】
| ・1st PCR反応液組成 | (μL) | |
| KOD One® PCR Master Mix | 20.6 | |
| 40μM S012 Primer | 0.3 | |
| 40μM S008 Primer | 0.3 | |
| cDNA溶液 | 20 | |
| Total Volume | 41.2 | |
・1st PCRサイクル
98℃ 3sec
↓
98℃ 10sec
55℃ 5sec
68℃ 2sec ×10
| ・2nd PCR反応液組成 | (μL) | |
| KOD One® PCR Master Mix | 10 | |
| 20μM S013 Primer | 0.5 | |
| 20μM S009 Primer | 0.5 | |
| 1st PCR溶液 | 3.4 | |
| 滅菌水 | 5.6 | |
| Total Volume | 20.0 | |
・2nd PCRサイクル
98℃ 3sec
↓
98℃ 10sec
61℃ 5sec
68℃ 2sec ×45
【NGSまでの処理方法】
PCR増幅産物をAgencourt® AMPure® XP beads (ベックマンコールター社)で精製し、10mM Tris-HCl(pH8.5)で溶出した。溶出した増幅産物をNextera® XT Index Kit v2(イルミナ社)にて処理し、再度、Agencourt® AMPure® XP beads で精製を行い、MiSeq®に供した。
<結果・考察>
得られたシーケンスリードから、亜系統の割合を計算した。
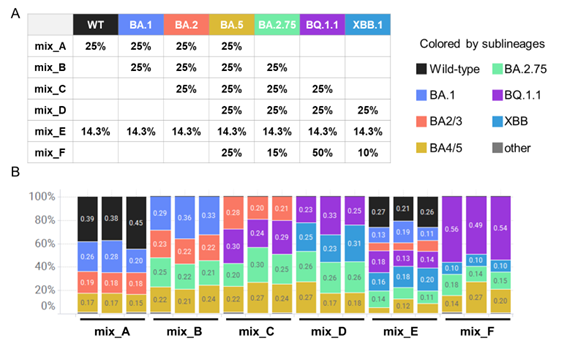







A : SARS-CoV-2亜系統の混合比率理論値
B : NGS結果から算出されたSARS-CoV-2亜系統の割合
KOD One®を使用したアンプリコンシーケンスの結果、理論値に近い亜系統の割合が算出されました。KOD One®は増幅バイアスが少なく、変異率も低いため、シーケンスターゲットの増幅にも最適であると考えます。
<東洋紡からのコメント>
今回紹介した研究は、下水疫学の調査に応用されています。
より詳細を確認なさりたい方は以下をご参照ください。
Targeted amplicon sequencing of wastewater samples for detecting SARS-CoV-2 variants with high sensitivity and resolution - PMC (nih.gov)
DOI: 10.1016/j.scitotenv.2023.164766
PCRによるシーケンスサンプルの調製は、バイアスを発生させる原因になりますので、増幅バイアスの少ない試薬を選択することが重要です。KOD One®は増幅バイアスが少なく、16S rRNAアンプリコンシーケンス(菌叢解析)などにも応用可能です。
高速かつ正確な増幅ができることから、ショートリードだけでなくロングリードのアンプリコン作製にも好評いただいております。