「源流を遡る」 第9回
第9回 パズル <2008年4月>
 前回は、1970年にSmithらによってⅡ型制限酵素が発見されたところまで報告しました。さらに調べると、この話にはさらに興味をそそられる続きがありましたので、今回はその調査結果を報告させていただこうと思います<今回は、お話というよりも、謎解きという形になりますが、ご了承ください>。
前回は、1970年にSmithらによってⅡ型制限酵素が発見されたところまで報告しました。さらに調べると、この話にはさらに興味をそそられる続きがありましたので、今回はその調査結果を報告させていただこうと思います<今回は、お話というよりも、謎解きという形になりますが、ご了承ください>。
制限酵素HindII(この論文の中では、”endonucelase R”と呼ばれています)を発見したSmithらは、この酵素によって切断されたDNA断片が特定の大きさを示すことなどから、この制限酵素はDNA中の特定の配列を認識して切断するのではないかと考えていました。しかし当時の技術力では、到底切断点付近をシーケンスすることなどできません。シーケンス法がSangerやGilbertらによって華々しく報告されるのは7年後の話です。
それでは、どのようにして切断点の配列を決定したのでしょう? それには、まさにパズルを解くような作業が必要でした。
| T.J. Kelly and H.O. Smith, A Restriction Enzyme from Hemophilus influenzae. II. Base sequence of the recognition site J. Mol. Biol., 51: 393-409 (1970) |
彼らは、まず、33Pでインターナルラベル(つまりすべてのDNAのリン酸結合に用いられているリン酸基を33Pでラベル)したT7ファージのDNAを調製しました。これがすべての実験に用いられる共通の鋳型となります。 次に彼らは、ラベルしたDNAをHindIIで切断し、その切断点の5’末端を今度は32Pで標識しました。このDNAは様々なヌクレアーゼを用いて消化されることになります。
次に彼らは、ラベルしたDNAをHindIIで切断し、その切断点の5’末端を今度は32Pで標識しました。このDNAは様々なヌクレアーゼを用いて消化されることになります。
2種類のラベルをした理由は以下のとおりです。最終的には、それぞれのヌクレオチドはThin-Layer Chromatography(TLC)で分離された後、X線フィルムを当ててその場所を特定し、同定することになります。そのとき、33Pはエネルギーが低く、直接X線フィルムを当てなくては検出できません。それに対して、32Pはエネルギーが強く途中に薄いフィルムを挟んで露光してもきちんと検出できるというメリットがあります。彼らはその性質に着目して2種類のラベル方法を使い分ける工夫を行ったのです。
上で簡単に述べましたが、当時、既に、すい臓由来のDNase Iやヘビ毒ホスホジエステラーゼなど、DNAを様々な様式で分解する種々の酵素の性質がかなり詳しく調べられていました。ここで次なる工夫です。彼らは、これらの酵素で限定分解した5’末端を含む2、3ヌクレオチドからなる断片について詳しく解析しました。つまり、「限定分解した断片を、さらに完全に分解し、33Pを指標として解析することで、それぞれの断片がどの塩基から構成されていたかが分かります」し、「32Pの解析をすることで、その断片の5’末端に含まれる塩基が分かる」という仕組みです。詳細に関しては割愛しますが、かなり工夫をこらしたテクニックが用いられていますので、興味のある方は是非論文をチェックされては如何かと思います。
さて、彼らの結果を簡単に下図にまとめてみました。皆さんは、先を読み進まずにこの図から、この酵素がどのような部位でDNAを切断するのかを推測してみてください。まさにパズルを解く要領です。[ヒント:この酵素は6塩基認識であり、その中にはPy (GC)とPu (AG)を大まかに認識するところも含まれます。この酵素は平滑末端を生じます]
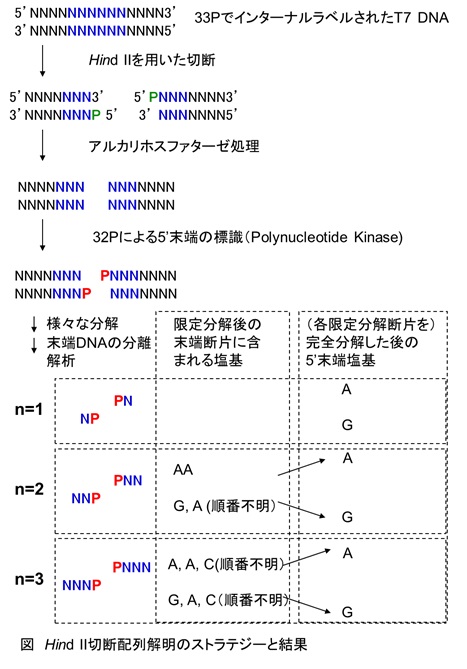
解説しますと、まず、n=1のところが既にAとGですので、切断点の5’末端はAもしくはG(すなわちPu)ということになります。この解析結果は、T7 DNAを用いて行われたもので、複数の断片の混合物のはずですから、上の結果ではAのものとGのものが混在した結果になっています。以前ご紹介した「人工生命(J.チャーファス著)」では、Smithらは、最初に得られたこの結果を見て少しがっかりしたというエピソードが書かれています。彼らとしては、特定の塩基のみを認識して欲しいという願望があったのでしょう。
では次の、n=2のところです。片方は5’AAで問題ありません。もう片方は、5’側がGなので、5’GAが正しいと考えられます。
続いてn=3ですが、ここまでくると簡単で、AACとGACです。
ここでは割愛しますが、n=4番目の解析により4番目の塩基はランダムだったようです。
まとめると、この酵素は、
5’ GTPy│PuAC 3’ (GTT│AAC or GTC│GAC)
3’ CAPu│PyTG 5’ (CAA│TTG or CAG│CTG)
とういうサイトを切断すると推定できます。
さらに彼らは、ヘビ毒ホスホジエステラーゼと脾臓ホスホジエステラーゼを用いる実験から切断後のリン酸の位置の確認も行いました(それぞれのホスホジエステラーゼはDNAを分解後、それぞれp-dNとdN-pを生じます)。切断後、リン酸基は以下のように配分されます。この制限酵素の性質は、例外はありますが、現在用いられているほぼすべて制限酵素に当てはまります。
5’ pGpTpPy│pPupApC 3’
3’ pCpApPup│pPypTpG 5’
今回ご紹介したこの論文と、前回ご紹介したHindIIを発見したという論文は同じジャーナルの同じ号に続けて紹介されています。今回私は、この両方の論文を読む機会があった訳ですが、私の持っている制限酵素に関する知識のかなりの部分が、この2報の論文に集約されているように感じました。制限酵素のバッファーの至適塩濃度やマグネシウム濃度の話、切断配列、切断後のリン酸基の位置などなど。また、使われている技術も、現在に通じるものばかりです。
さらに今回、Smithらの巧みな実験手法を知って「目からうろこ」が落ちる思いでした。今回ご紹介した配列決定は、現在の教科書からの知識や技術力をもってすれば、学生さんでも2,3日でできるかも知れません。しかし、シーケンサーを取り上げてしまえば、まったく手も足も出ないに違いありません。
次号は、1977年に報告された画期的な技術に遡るつもりです。では。
(T.K.)
2008年4月掲載
 Indexに戻る
Indexに戻る







