実施例
KOD One 実施例14 遺伝子の発現ベクター間の移し替え
データご提供 : 慶應義塾大学 理工学部 生命分子工学研究室 藤原 慶 様
<実験の目的>
別々にクローン化した遺伝子を、別の発現ベクターに移し替える。
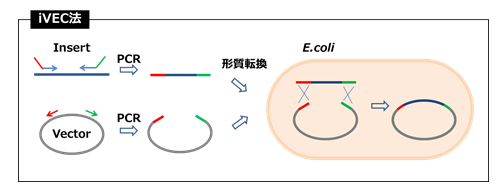 <実験方法>
<実験方法>
【サンプルの種類】
プラスミド DNA
【サンプルの調製方法】
キットで精製したDNA
【ターゲット遺伝子名と長さ】
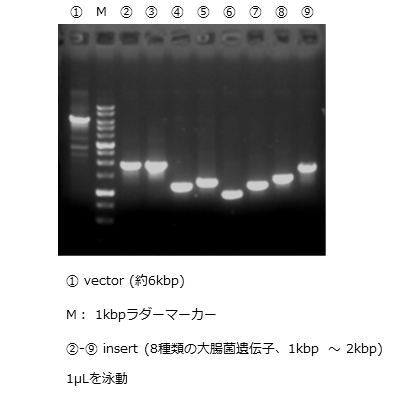
ベクターA(pET15由来) 6,050bp 大腸菌遺伝子A-H 1,000~2,000bp
| 【プライマー配列】 | |
| ベクター用 Forward Primer : 長さ : 20nt Tm : 55.4℃ Reverse Primer : 長さ : 18nt Tm : 56.9℃ |
遺伝子用 Forward Primer : 長さ : 23nt Tm : 58.8℃ Reverse Primer : 長さ : 21nt Tm : 53.8℃ |
【相同領域】
(今回の実験では、移し替えるplasmid自体に相同配列があるため、相同領域にプライマー配列が含まれます)
Forward側 : 81nt Reverse側 : 117nt
【反応液組成】 【PCRサイクル】
| (μL) | ||||||
| 滅菌蒸留水 | 1.4 | 98℃ 1min. | ||||
| KOD One® PCR Master Mix | 2.5 | 96℃ 10sec. | 25cycles | |||
| 3μM Primer F | 0.5 | 55℃ 10sec. | ||||
| 3μM Primer R | 0.5 | 72℃ 10-35sec. | ||||
| 50ng/μL Template | 0.1 | |||||
| Total Volume | 5.0 | |||||
【測定機器】ChemiDoc MP (Bio-Rad)
PCR後、反応液 5μL に対しDpnI(NEB)を0.1μL添加
↓
 37℃ 40分(この間に電気泳動)
37℃ 40分(この間に電気泳動)
↓
Vector 1.5μL, 遺伝子 1μLを混合し、12μLの iVEC3株のコンピテントセルと混合
(資源研より供与された株を凍結保存プロトコールによりコンピテントセル化したものを使用)
↓
On ice 20分
↓
LB培地 400μLを添加し、37℃で60分静置
↓
遠心(12,000xg 30秒)により菌体を回収し、上清を捨てる
↓
10μLの10mM Tris-HCl(pH8.0)で懸濁し、即座にプレートへまく
↓
翌日、生えたコロニーに対するコロニーPCRもKOD Oneで行った(1サンプルあたり合計3μLの系)
<結果・考察>
各遺伝子につき1コロニーをつついた。8コロニー中7コロニーが当たり
<感想・ご意見等>
他社の高速PCR酵素から切り替えたところ、ベクターの増幅効率が倍以上になり、なおかつベクターPCRの失敗がほとんどなくなりました。また、iVEC3法での形質転換を活用することで、PCRからプレーティングまで2時間で終えることができるようになりました。
 KOD One 製品ページ
KOD One 製品ページ
 実施例一覧ページ
実施例一覧ページ







